Локус количественных признаков
Локусы количественных признаков, сокращённо — ЛКП (от англ. Quantitative Trait Loci — QTLs), являются участками ДНК, либо содержащими гены, либо сцепленными с генами, которые отвечают за тот или иной количественный признак. Количественные признаки относятся к характеристикам, которые различаются по степени своего выражения и могут быть отнесены к полигенным эффектам, то есть являются продуктом двух или более генов.
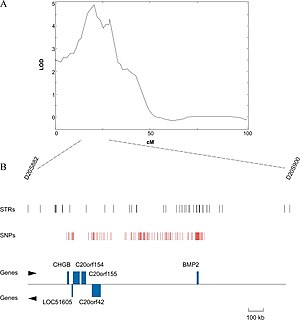

Природа количественных признаков править
Полигенное наследование править
Для количественных признаков характерно полигенное наследование, также известное как «множественное», или мультифакторное. Оно относится к наследованию характеристик фенотипа, за которые отвечают два или более гена, или взаимодействие последних с окружающей средой, или и то, и другое. В отличие от моногенных признаков, полигенные признаки не подчиняются законам Менделя. Вместо этого фенотипические признаки обычно варьируют с равномерным отклонением, изображаемым при помощи кривой нормального распределения[1].
Примером полигенных признаков может служить цвет человеческой кожи. За определение естественного цвета кожи индивида отвечают многие гены, так что изменение лишь одного из них едва ли приведёт к существенным переменам в цвете.
Полигенные заболевания править
Многие наследственные заболевания имеют полигенную природу; к таковым относятся аутизм, рак, диабет и другие. Большинство фенотипических характеристик являются результатом взаимодействия множества генов.
Примеры заболеваний мультифакторной этиологии:
- Врожденные пороки
- Расщепление нёба[2][3].
- Дисплазия тазобедренного сустава.
- Врождённые пороки сердца.
- Дефект нервной трубки.
- Пилоростеноз (сужение привратника желудка).
- Деформации стопы.
- Болезни, развивающиеся у взрослых
- Сахарный диабет[3].
- Рак[3].
- Эпилепсия.
- Глаукома.
- Артериальная гипертензия.
- Ишемическая болезнь сердца.
- Биполярное аффективное расстройство.
- Шизофрения.
Считается, что заболевания мультифакторной природы составляют большинство среди генетических нарушений человека[4].
Размеры ЛКП править
Показано, что комплексная генетическая система, включающая группу полигенов и образующая локус количественных признаков, может быть достаточно чётко локализованной (компактной). Предполагается, что ЛКП существует в геномах многих высших организмов как цельная единица, и, как было оценено в 1980 году, размер ЛКП может составлять у человека 20 сантиморганид[5].
QTL-картирование править
Картирование участков генома, включающих гены, связанные с определённым количественным признаком, производится при помощи молекулярных маркеров, таких как AFLP (полиморфизм длин амплифицированных фрагментов (в сайтах рестрикции), или ПДАФ), микросателлитов или SNP (однонуклеотидный полиморфизм). Это первый шаг в идентификации и определении генов, отвечающих за изменчивость признаков. Затем проводят анализ последовательности ДНК и определяют кандидатные гены, которые могут участвовать в контроле изучаемого количественного признака. В тех случаях, когда геном организма ещё не секвенирован полностью, прибегают к позиционному клонированию. На основе данных сцепления количественного признака с генетически локализоваными полиморфными маркерами выделяют геномные клоны, соответствующие картированному локусу, и путём их секвенирования идентифицируют вероятные гены, связанные с проявлением признака[6][7][8].
Значение в сельском хозяйстве править
У сельскохозяйственных растений и животных множество хозяйственно полезных признаков (продуктивность, выход рисовой крупы, качество яиц и т. п.) наследуются по сложному полигенному типу и находятся под контролем многих генов, расположенных в QTL-локусах[9][10]. Сведения о нуклеотидных последовательностях из районов QTL могут быть использованы, например, в практическом животноводстве для проведения селекции посредством молекулярных маркеров (англ. marker-assisted selection — MAS)[8]. Исследование комплексной молекулярной архитектуры этих локусов является важным и с точки зрения общей генетики. При этом часто применяют методику позиционного клонирования районов хромосом, контролирующих количественные признаки, которые предварительно локализованы с помощью полиморфных (например, микросателлитных) маркеров[11].
См. также править
Примечания править
- ↑ Lewis R. Multifactorial Traits // Human Genetics: Concepts and Applications. — 5th edn. — New York, NY, USA: McGraw-Hill, 2003. — 454 p. — ISBN 0071198490. Архивировано 1 апреля 2015 года. (англ.) (Дата обращения: 18 октября 2020) Архивированная копия. Дата обращения: 7 октября 2020. Архивировано 9 июля 2019 года.
- ↑ Proud V., Roberts H. Multifactorial Inheritance (англ.). Health Library: Health Information: Medical Genetics. Norfolk, VA, USA: Children's Hospital of The King's Daughters (31 декабря 2005). Дата обращения: 6 января 2007. Архивировано 15 октября 2006 года.
- ↑ 1 2 3 Multifactorial Inheritance (англ.). The March of Dimes: Pregnancy and Newborn Health Education Centre. White Plains, NY, USA: March of Dimes Foundation. Дата обращения: 6 января 2007. Архивировано 10 сентября 2010 года.
- ↑ Tissot R. Course Director E. Kaufman: Multifactorial Inheritance (англ.). Human Genetics for 1st Year Students. Chicago, IL, USA: Department of Molecular Genetics, College of Medicine, University of Illinois at Chicago. Дата обращения: 19 марта 2015. Архивировано 3 марта 2016 года.
- ↑ Арефьев В. А., Лисовенко Л. А. Локусы количественных признаков // Биология: Молекулярная биология и генетика. Толковый словарь. — 1995.
- ↑ Александров А. А., Ковалёв П. В. Позиционное клонирование. База знаний по молекулярной и общей биологии человека. М.: HUMBIO; ООО «Лайт Телеком». Дата обращения: 18 октября 2020. Архивировано 22 февраля 2020 года.
- ↑ Тарантул B. З. Позиционное клонирование, позиционное картирование (positional cloning, positional mapping) // Толковый биотехнологический словарь. Русско-английский. — M.: Языки славянских культур, 2009. — 936 с. — ISBN 978-5-9551-0342-6. Архивировано 20 марта 2015 года. (Дата обращения: 18 октября 2020) Архивированная копия. Дата обращения: 22 марта 2015. Архивировано 23 февраля 2020 года.
- ↑ 1 2 Сазанов А. А., Царёва В. А., Смирнов А. Ф., Вардецка Б., Корчак М., Ящак К., Романов M. Н. (2003-04-03). "Позиционное клонирование районов хромосом, контролирующих количественные признаки у кур" (PDF). Тез. докл. Итоговый семинар по физике и астрономии по результатам конкурса грантов 2002 г. для молодых учёных Санкт-Петербурга (PDF). СПб.: Физико-технический институт им. А. Ф. Иоффе. pp. 44—45. Архивировано из оригинала (PDF) 2 апреля 2015. Дата обращения: 18 октября 2020.
{{cite conference}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Song B.-K., Nadarajah K., Romanov M. N. Ratnam W. Cross-species bacterial artificial chromosome (BAC) library screening via overgo-based hybridization and BAC-contig mapping of a yield enhancement quantitative trait locus (QTL) yld1.1 in the Malaysian wild rice Oryza rufipogon (англ.) // Cellular & Molecular Biology Letters : журнал. — Wrocław, Poland; Berlin, Heidelberg, Germany: Cellular & Molecular Biology Letters, University of Wrocław, Ministry of Science and Higher Education, Poland; Springer Science+Business Media, 2005. — Vol. 10, no. 3. — P. 425—437. — ISSN 1425-8153. — PMID 16217554. Архивировано 20 марта 2009 года. (Дата обращения: 18 октября 2020)
- ↑ Сазанов А. А., Царёва В. А., Смирнов А. Ф., Вардецка Б., Корчак М., Ящак К., Романов M. Н. (2004-04-26). "Позиционное клонирование локусов количественных признаков у домашней курицы" (PDF). Тез. докл. Итоговый семинар по физике и астрономии по результатам конкурса грантов 2003 года для молодых учёных Санкт-Петербурга (PDF). СПб.: Физико-технический институт им. А. Ф. Иоффе. p. 42. Архивировано из оригинала (PDF) 2 апреля 2015. Дата обращения: 18 октября 2020.
{{cite conference}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Sazanov A. A., Romanov M. N., Wardecka B., Sazanova A. L., Korczak M., Stekol'nikova V. A., Kozyreva A. A., Smirnov A. F., Dodgson J. B., Jaszczak K. (2004-07-06). "Chromosomal localization of GGA4 BACs containing QTL-linked microsatellites". In Coordinator H. Hayes (ed.). Cytogenetic and Genome Research. Abstracts of the 16th ECACGM (European Colloquium on Animal Cytogenetics and Gene Mapping), Jouy-en-Josas, July 6—9, 2004. Vol. 106 (1). Basel: Karger Publishers. p. 19. doi:10.1159/000078555. OCLC 254439250. Abstract P18. Дата обращения: 18 октября 2020.
{{cite conference}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) (англ.) Архивированная копия. Дата обращения: 18 октября 2020. Архивировано 12 июня 2018 года.