Пиросеквенирование
Пиросеквени́рование — это метод секвенирования ДНК (определение последовательности нуклеотидов в молекуле ДНК), основанный на принципе «секвенирование путём синтеза». При включении нуклеотида происходит детекция высвобождающихся пирофосфатов[1]. Технология была разработана Полом Ниреном[англ.] (швед. Pål Nyrén) и его студентом Мустафой Ронаги[англ.] англ. Mostafa Ronaghi) в Королевском технологическом институте (Стокгольм) в 1996 году[2][3][4].
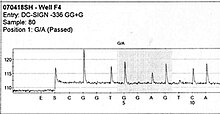
Процедура
правитьИдея пиросеквенирования заключается в регистрации пирофосфата, который образуется при присоединении очередного нуклеотида ДНК-полимеразой. Детекция пирофосфата осуществляется за счёт каскада химических реакций, который заканчивается выделением кванта света[5].
Прежде всего создаётся иммобилизованная на твёрдой фазе клональная библиотека[англ.] одноцепочечных фрагментов ДНК (например, при помощи мостиковой полимеразной цепной реакции, ПЦР). Ко всем фрагментам ДНК присоединяется адаптер, с которым будет комплементарно связываться праймер — затравка для синтеза комплементарной цепи ДНК-полимеразой. Далее производится серия последовательных циклов, в процессе которых к закреплённой на твёрдой фазе ДНК по очереди добавляют дезоксинуклеотидтрифосфаты всех четырёх типов: A, T, G, C. Если на секвенируемой цепи ДНК есть комплементарный к добавленному нуклеотид, то при образовании фосфодиэфирной связи побочным продуктом станет пирофосфат. Он активирует каскад химических реакций: пирофосфат вместе с аденозинсульфофосфатом (АСФ) при помощи фермента АТФ-сульфурилазы[англ.] образуют АТФ, который является источником энергии для проведения реакции окисления люциферина в оксилюциферин с выделением кванта света. Интенсивность выделяемого света пропорциональна числу включённых в цепь нуклеотидов (чем больше подряд одинаковых нуклеотидов, тем сильнее световой сигнал). Детекция света осуществляется ПЗС-матрицей и анализируется с помощью программного обеспечения, которое строит по пирограмме последовательность нуклеотидов. Нуклеотиды, не вовлечённые в синтез новой цепи, а также АТФ разрушаются ферментом апиразой[англ.]. После этого начинается следующий цикл, то есть добавляется нуклеотид другого типа[6].
Позднее Маргулис и соавторы предложили сочетание пиросеквенирования с эмульсионной ПЦР[7].
Коммерциализация
правитьКомпания Pyrosequencing AB, которая располагается в Уппсале, Швеция, коммерциализовала технологию и реагенты для секвенирования коротких участков ДНК. В 2003 году Pyrosequencing AB переименована в «Biotage». В 2008 году Qiagen приобрел Biotage[8]. На принципе пиросеквенирования основана коммерческая технология 454 Life Sciences (компания Roche Applied Science[англ.])[7].
Достоинства и недостатки
правитьПо сравнению с секвенированием по Сэнгеру, пиросеквенирование выгодно отличается стоимостью, а также тем, что за один раз можно получить сотни тысяч прочтений[англ.]. Однако пиросеквенирование имеет и ряд недостатков. Так, с помощью этого метода невозможно безошибочно секвенировать протяжённые участки, состоящие из одного и того же нуклеотида. Кроме того, оно позволяет получать лишь прочтения небольшой длины. Четвёртое поколение технологий пиросеквенирования, например, GS FLX Titanium, позволяет получать прочтения длиной более 400 пар оснований[9].
Литература
править- Д. В. Ребриков, Д. О. Коростин, Е. С. Шубина, В. В. Ильинский. NGS: высокопроизводительное секвенирование / под общей редакцией Д. В. Ребрикова. — М.: БИНОМ. Лаборатория знаний, 2015. — 232 с. — ISBN 978-5-9963-0373-1.
Примечания
править- ↑ Definition of pyrosequencing from the Nature Reviews Genetics Glossary. Дата обращения: 28 октября 2008. Архивировано 30 марта 2012 года.
- ↑ Ronaghi M., Uhlén M., Nyrén P. A sequencing method based on real-time pyrophosphate. (англ.) // Science (New York, N.Y.). — 1998. — Vol. 281, no. 5375. — P. 363—365. — PMID 9705713.
- ↑ Ronaghi M., Karamohamed S., Pettersson B., Uhlén M., Nyrén P. Real-time DNA sequencing using detection of pyrophosphate release. (англ.) // Analytical biochemistry. — 1996. — Vol. 242, no. 1. — P. 84—89. — doi:10.1006/abio.1996.0432. — PMID 8923969.
- ↑ Nyrén P. The history of pyrosequencing. (англ.) // Methods in molecular biology (Clifton, N.J.). — 2007. — Vol. 373. — P. 1—14. — doi:10.1385/1-59745-377-3:1. — PMID 17185753.
- ↑ Ребриков и др., 2015, с. 28.
- ↑ Ребриков и др., 2015, с. 28—30.
- ↑ 1 2 Ребриков и др., 2015, с. 30.
- ↑ Qiagen acquires biosystems business from Biotage. Qiagen press release, October 1st, 2008. Дата обращения: 3 мая 2009. Архивировано 15 июля 2011 года.
- ↑ Siqueira J. F. Jr., Fouad A. F., Rôças I. N. Pyrosequencing as a tool for better understanding of human microbiomes. (англ.) // Journal of oral microbiology. — 2012. — Vol. 4. — doi:10.3402/jom.v4i0.10743. — PMID 22279602.
Ссылки
править- Натальин, Павел. 454-секвенирование (высокопроизводительное пиросеквенирование ДНК). // Сайт Biomolecula.ru (6 февраля 2008). Дата обращения: 21 марта 2018. Архивировано 7 апреля 2018 года.
- Недолужко, Артём; Пташник, Ольга. 12 методов в картинках: секвенирование нуклеиновых кислот. // Сайт Biomolecula.ru (11 августа 2017). Дата обращения: 21 марта 2018.
Эта статья входит в число добротных статей русскоязычного раздела Википедии. |